Presentación de Casos y Revisión de Temas
YENNY MAGNOLIA MONTENEGRO MEDINA*, ADONIS TUPAC RAMÍREZ CUÉLLAR**,CARLOS MARIO MUÑETÓN**
Resumen
El 15% de los CCR, está asociado con un patrón de herencia autosómico dominante y se estima que aproximadamente 12.940 nuevos casos y 6.580 definiciones tienen antecedentes familiares. Por lo tanto, el factor genético atribuido a su etiología se considera cada vez de una mayor importancia. Gracias a los grandes avances de la biología molecular durante la pasada década, se propuso la pérdida de heterocigocidad y la presencia de mutaciones germinales en el sistema de reparación del ADN como mecanismos que predisponen al CCR. La Poliposis Adenomatósica Familiar (FAP) causada por el primer mecanismo se presenta en menos del 1% de todos los casos de CCR, mientras que el segundo mecanismo (CCNPH) ocasiona entre el 10 y 15%.
Introducción
En Estados Unidos, el cáncer colo-rectal (CCR) se considera la segunda causa de muerte por cáncer; en 1999 se informaron 94.700 nuevos casos de cáncer de colon y 34.700 de recto y durante este mismo periodo se informaron 56.400 muertes por esta patología, de las cuales el 10% (12.940 nuevos casos y 5.660 defunciones) se atribuyó a factores genéticos como en el Cáncer Colo-rectal Heredado sin Poliposis (CCHSP) y la Poliposis Adenomatósica Familiar (PAF). El CCHSP, es una entidad autosómica dominante que causa entre el 5 y 15% de todos los cánceres colo-rectales. Esta patología es causada por mutaciones en la línea germinal de uno de los cinco genes de reparación (MLH1, MSH2, PMS1, PMS2 y MSH6) de los cuales, los afectados con mayor frecuencia son MLH1 y MSH2, aunque la inactivación de cualquiera de ellos produce inestabilidad genómica (inserción o deleción de secuencias de microsatélites); esta, finalmente, conduce a la acumulación de mutaciones somáticas importantes en la iniciación y progresión de la neoplasia1-10. La presencia de mutaciones en estos genes, tanto en los pacientes afectados como en sus familiares es de gran importancia, dado que la mutación está directamente relacionada con el fenotipo de la enfermedad y la resistencia a ciertas drogas antineoplásicas, por lo cual en algunos centros del mundo se ha implementado la identificación de portadores en familias afectadas, detectando un riesgo de desarrollar cáncer antes de los 75 años, 80 veces mayor en estos individuos comparado con la población general. La PAF, causa entre el 1 y 5 de los cánceres colorectales heredados y clínicamente es una entidad de fácil diagnostico. Molecularmente se caracteriza por grandes deleciones sobre oncogenes y genes supresores de tumor que finalmente conllevan al desarrollo de la enfermedad
Epidemiología
Durante varios años, el cáncer colo-rectal (CCR) ha sido la cuarta neoplasia más frecuente en el mundo y la segunda causa de muerte por cáncer en los Estados Unidos, con un promedio de 136.000 nuevos casos y 55.000 muertes por año1,2. En 1999, en los Estados Unidos, se informaron 94.700 nuevos casos de cáncer de colon y 34.700 de cáncer de recto, lo cual representa el 15% de todas las neoplasias en humanos. La mortalidad durante este mismo periodo fue de 47.900 casos de cáncer de colon y de 8.700 de cáncer de recto; convirtiéndose así en uno de los principales problemas de salud pública. Sólo en 1999 se reportaron en el mundo 875.000 nuevos casos de CCR3. La variación geográfica de la incidencia del CCR en el mundo es muy alta, siendo Nueva Zelanda e Irlanda los países más afectados, seguidos por los Estados Unidos; la más baja se presenta en la India. El 15% de los CCR, está asociado con un patrón de herencia autosómico dominante y se estima que aproximadamente 6.580 casos tienen antecedentes familiares; por lo tanto, el factor genético atribuido a su etiología se considera cada vez de mayor importancia. Gracias a los grandes avances de la biología molecular durante la pasada década, se propuso la pérdida de heterocigocidad y la presencia de mutaciones germinales en el sistema de reparación del ADN como mecanismos que predisponen al CCR. La Poliposis Adenomatósica Familiar (PAF) causada por el primer mecanismo se presenta en menos del 1% de todos los casos de CCR, mientras que el CCHSP es producido por el segundo mecanismo y ocasiona mas del 5%2-5.
Predisposición Genética
El CCR heredado se clasifican en dos grupos: los síndromes polipoides y los no polipoides, sobre los cuales adelante se hace mayor énfasis2-7.
Poliposis Adenomatósica Familiar (PAF)
Descrita hace más de cien años, se caracteriza clínicamente porque los pacientes tienen más de 100, y hasta 1.000, pólipos adenomatósicos colónicos que empiezan a aparecer entre los 10 y 20 años de edad, inicialmente en la región rectosigmoidea del colon, y con frecuencia producen hemorragia rectal. Esta patología se hereda de forma autosómica dominante con penetrancia completa, aunque existen casos esporádicos sin historia familiar que parecen haber presentado mutaciones espontáneas. La PAF generalmente se manifiesta entre los 50 y 60 años de edad con alto riesgo de desarrollar otros carcinomas en tiroides, estómago, cerebro y sarcomas óseos5,6,9-11.
Genotípica y fenotípicamente las familias con PAF son muy heterogéneas pero generalmente se caracterizan por presentar aneuploidias y mutaciones específicas en los genes APC, k-ras, y p53. Existen algunas variantes de PAF como:
A. El Síndrome de Gardner, donde las manifestaciones extracolónicas principales son osteomas y fibromas en la piel.
B. El Síndrome de Peutz-Jegher asociado a geno-dermatosis y múltiples melanosis mucocutáneos en la cavidad oral; su gen, fue recientemente mapeado en el cromosoma 19p 13.36.
C. La Poliposis Adenomatósica Familiar Atenuada (PAFA), la cual se caracteriza por el desarrollo de un menor número de adenomas colónicos con predilección por la región proximal del colon; se presenta a mayor edad que la PAF clásica9.
El fenotipo de la enfermedad parece estar relacionado con la localización de la mutación sobre el gen APC así: en PAFA, la mutación frecuentemente se encuentra en la región 5 entre los codones 1285 y 1465, mientras que la aparición de hipertrofias congénitas del pigmento epitelial de la retina en el Síndrome de Gardner se asocia con mutaciones en los codones 542 a 1809 y la presencia severa de metástasis extracolónicas con cambios en los codones 1465 a 1546 y 2621. El tratamiento para estos individuos es proctocolectomia o colectomia subtotal con un seguimiento del remanente rectal por rectosig-moidoscopia anual5,6.
Cáncer Colo-rectal No Polipoide Hereditario (CCNPH)
En 1821 Warthin6 describió diferentes familias con cáncer, entre ellas ” La familia G”, caracterizada por presentar varios casos de cáncer de estómago y útero en las primeras generaciones y en las siguientes una mayor incidencia de CCR. En 1971 Lynch y Krush lo denominaron como Síndrome de Lynch o CCNPH4-6.
Básicamente el Síndrome de Lynch se caracteriza por: a) tener un patrón de herencia autosómica dominante, b) penetrancia del 85 al 90% y c) diagnóstico de CCR antes de los 45 años de edad. Histopatológicamente sus características son, la diferenciación e incremento de los marcadores celulares, infiltración linfocítica peritumoral mezclada con células tumorales que aumenta el riesgo de hacer metástasis a sitios como endometrio, ovario, estómago, tracto hepatobiliar, uretra y pelvis renal. Adicionalmente en algunas familias con CCNPH se ha observado un elevado porcentaje de cáncer de seno5,7,9. Sin embargo, para aumentar la sensibilidad y especificidad en la definición del diagnóstico de CCNPH, se han definido los criterios de Amsterdam (ver tabla 1).

Molecularmente se observa inestabilidad de microsatélites (MIN), diploidia y mutaciones en los genes de reparación, del receptor del factor transfor-mante de crecimiento b tipo II y BAX. Al parecer los pacientes con mutación en hMSH2 tienen mayor probabilidad de desarrollar cáncer de endometrio y adenocarcinomas de estómago y ovario al compararlos con el hMLH12,6,7 ,12.
Genes Asociados a CCR
Gen de la Poliposis Adenomatósica Familiar (PAF)
Esta ubicado en el cromosoma 5q, codifica para una proteína citoplasmatica de 2.843 aminoácidos que se localiza en los tejidos adultos, y tiene función desco-nocida, aunque se ha observado que la región C-terminal puede unirse a los microtubulos y la región N-terminal está involucrada con la unión a las catenina a y b1,13. La frecuencia de mutaciones en el gen APC para la población general es de 1/1000 con penetrancia del 100%. Las mutaciones observadas incluyen desde grandes delecio-nes, hasta mutaciones puntuales e hipermetilaciones que afectan los mecanismos de transcripción y “splicing”. Sin embargo, más del 90% de los cambios descritos hasta el momento bloquean la producción de la proteína al formar codones de parada previos, lo que facilita un poco el diagnóstico por técnicas moleculares. Los datos sugieren que ambos alelos de APC deben estar mutados para que se presente el fenotipo, generalmente uno de forma heredada y el otro alelo durante divisiones poscigoticas o los dos alelos por mutaciones somáticos (cuando el tumor es esporádico)4.
Proteína del Sarcoma de Rous (RAS)
La principal vía de señalización mitógena en muchos tipos de células es la activación del gen ras. Tres genes diferentes de ras han sido identificados, H-ras sobre el cromosoma 11p, K-ras sobre 12q y N-ras sobre 1p; en términos bioquímicos son muy similares y todos pueden convertirse en oncogenes. Estos genes codifican para proteínas que se unen a secuencias ricas en guanina y controlan algunos eventos celulares como la formación del citoesqueleto de actina, el transporte de vesículas entre compartimentos y presumiblemente juega un papel importante en el control del ciclo celular a partir de los receptores de superficie13,14. En humanos únicamente se han hallado mutaciones en los codones 12,13 y 61 de K-ras, los cuales al parecer producen alteraciones en la actividad de la proteína.
Gen de la Deleción de Cáncer de Colon (DCC)
La pérdida de una copia del gen DCC que se localiza en el cromosoma 18q21 ha sido demostrada en más del 70% de los CCR y en el 50% de los adenomas. Este gen codifica para una proteína de 190 kd con estructura similar a la de un receptor de superficie celular. Ésta proteína se encuentra adherido a membranas y presenta particular homología con las moléculas de adhesión neuronal N-CAM. Su función esta asociada con la interacción entre células – matriz y/o con procesos de diferenciación15.
Genes de la Reparación Metil – Dirigida de Bases Mal Apareadas (mismatch)
El ADN es altamente reactivo y puede ser el blanco de numerosos agentes biológicos, físicos y químicos; sin embargo, es muy estable en su capacidad para conservar la información genética que contiene; dicha estabilidad puede atribuirse a su estructura de doble hélice y a la existencia de varios mecanismos de reparación en la molécula. La importancia de estos mecanismos radica en que una simple sustitución de una base en un gen puede modificar la función de la proteína corres-pondiente, causando alteraciones incluso letales16. Múltiples sistemas protegen el genoma de los mamíferos, desde la apoptósis que elimina células anormales de la población celular hasta las enzimas específicas de reparación que corrigen daños puntuales en el ADN, los cuales pueden clasificarse así:16
A. Reparación directa:
Monomerización del dimero de pirimidinas
Eliminación del grupo metilo de la 06 metil guanina
Monomerización del fotoproducto esporádico
B. Reparación por escisión
Reparación por escisión de bases
Reparación por escisión de oligonucleótidos
Reparación metil-dirigida de bases mal apareadas
(Mismatch repair)
C. Reparación Recombinacional o posreplicativa
D. Reparación SOS
El sistema de reparación metil dirigida de bases mal apareadas (mismatch Repair “MR”), tiene la capacidad de reconocer y reparar pares de nucleótidos lesionados por métodos físicos, incorporación errónea por la ADN polimerasa o heteroduplex formados durante eventos de recombinación genética. Esta incorporación incorrecta puede suceder durante la síntesis o en el ADN ya sintetizado; en el primer caso, la ADN polimerasa incorpora una base diferente a la indicada, mientras que el segundo puede ocurrir por des-aminación de la 5-metil citosina o adenina que conducen a la formación, respectivamente, de uracilo, timina e hipoxantina (16,20). Cuando la lesión escapa a estos mecanismos, actúan los genes del sistema de reparación de mismatch (MLH1, MSH2, MSH3 Y MSH6), los cuales requieren que la cadena a corregir no esté aún modificada por la metilación de la adenina localizada en secuencias GATC. El ATP, la proteína SSB (proteína que se une a ADN de una sola cadena) y los productos de los genes involucrados son indispensables para que haya una corrección eficiente. La proteína SSB se une fuerte y cooperativamente al ADN de cadena sencilla y además estimula la actividad de las ADN polimerasas II y III. La proteína hMSH2 junto con MSH3 ó MSH6, se une al ADN sobre el par de bases apareadas erróneamente y el gen hMLH1 actúa como una endonucleasa sitio específica, que rompe un enlace fosfodiester en el lado 5’ de la secuencia GATC no metilada. Se desconoce el papel específico del las otras proteínas20,21.
La expresión del gen hMLH1 ubicado en 3p21 es esencial para el mantenimiento de la estabilidad de los microsatélites y el funcionamiento del sistema de reparación que además de iniciar todo el proceso, aparentemente interviene en la regulación de los punto de control en las fases G1, M y G1 del ciclo celular, la segregación y formación de quiasmas durante la meiosis. Después de que hMLH1 da origen a las incisiones en el ADN que contiene la base mal apareada y no metilada, el gen uvrB elimina el fragmento abriendo un espacio de una kilobase como mínimo; posteriormente, la ADN polimerasa llena dicho espacio polimerizando los nucleótidos correctos para finalmente dejar actuar a la ADN ligasa16,20,22.
Genética Molecular del Cáncer Colo-Rectal CCR
Se conocen dos formas de CCR; una con predisposición hereditaria, de tipo autosómico dominante y otra completamente espontánea, que pone de manifiesto la interacción genética con los factores medioambientales en la etiología de este tumor. La mayoría de células neoplàsicas observadas en el CCR, comparte un alto grado de inestabilidad genómica producida por dos vías moleculares; la vía supresora y la vía mutadora; en la primera se observa inestabilidad cromosómica, mientras que en la segunda hay inestabilidad microsatélital. Algunas otras vías se han propuesto, como la del gen receptor de estrógenos y la de los genes relacionados con los puntos de control de la mitosis; sin embargo, estas últimas no han sido demostradas4.
Vía Supresora
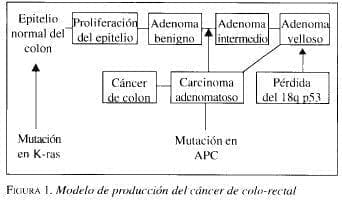
Se presenta en el 80% de los casos de CCR esporádico y en la mayoría de PAF. La inestabilidad cromosómica se caracteriza por el desarrollo de tumores con aneuploidías, pérdida de heterocigosidad en múltiples loci y acumulación de mutaciones en los genes reguladores de genes supresores o que activan oncogenes como APC, p53 y ras5,12,18,19,23. Este modelo se planteó al observar el grado de transformación de las células del epitelio intestinal hacia células tumorales. La primera característica es que requieren mutaciones en cuatro o cinco genes específicos para que se produzca el crecimiento maligno y en segundo lugar que el orden de las mutaciones aunque no es estricto, generalmente se presenta como se menciona adelante; sin embargo, lo importante, más que el orden, es la acumulación de mutaciones24. La primera mutación se localiza en el gen APC, convirtiendo el epitelio normal en uno o más tumores benignos (adenomas) por pérdida en el control de la división celular, adhesión y migración. Esta vía envuelve las cateninas b y el factor de células T, dando señales de proliferación transcripcional que pueden operar por la vía de C myc . En casos espontáneos, la mutación ocurre en una sola célula, por lo cual el adenoma resultante, es un clon celular. La siguiente mutación es en el oncogen k-ras y la acumulación de estas dos mutaciones en los adenomas estimula su crecimiento. Finalmente, la mutación en p53 causa la transición a células cancerosas porque no hay regulación del ciclo celular para el paso de G1 a S. Otros cambios muy comunes incluyen la pérdida de copias de genes DPC4/Smad4 o DCC localizado en el cromosoma 18 y del gen Peutz-Jegher situado en el cromosoma 19 muy característicos en los tumores esporádicos4. El proceso de metástasis ocurre después de la formación de las células cancerosas e incluye un número desconocido de mutaciones puntuales (ver figura 1).
Vía Mutadora
Este mecanismo se presenta en CCNPH, enfermedad que constituye 10 a 15% de los casos de CCR. Estos tumores se caracterizan por presentar poliploidia o pseudopoliploidia. Además, se observa ausencia de mutación en los genes tumorales comunes y acumu-lación de numerosas mutaciones en microsatélites constituidos principalmente por polidesoxiadenosinas. La acumulación de estas mutaciones tiene consecuencias significativas para la estructura y función del genoma; ellas pueden alterar la organización de la cromatina e inducir cambios en la estructura del nucleosoma o modificar el patrón de expresión génica por mutaciones en los potenciadores y/o promotores de los genes. El momento en que ocurre la inestabilidad se desconoce y tampoco se sabe si el defecto continúa durante el desarrollo del tumor; no obstante, se ha observado que las mutaciones son homogéneas en el mismo tumor pero difieren entre los tumores. Lo anterior sugiere que la vía Mutadora continúa después de la replicación6,9,24.
La gran inestabilidad genómica en los microsatélites se explicó al comprobar la presencia de daño en los genes de reparación del ADN a las cuales, Larry Loeb denominó Mutaciones del Fenotipo Mutador (MFM). Las MFM identificadas se localizan en los cromosomas 2q15-16 y 3p21, donde se ubican los genes hMSH2 y hMLH1, con los cuales se planteó el carácter recesivo del fenotipo Mutador .
La alteración en estos dos genes a su vez induce nuevas mutaciones como la inactivación del gen BAX que regula la apoptosis. En el 50% de los tumores de colon se observan otras mutaciones similares en genes como el TGFbRII, hMSH3 y hMSH6, estos dos últimos también asociados a la reparación del ADN4,25.
La presencia de deleciones o inserciones que alteran el marco de lectura de los aminoácidos en hMSH3 y hMSH6 se debe a la presencia de mutaciones sobre sus microsatélites de desoxiadenina, las cuales se producen por un gen mutador que muta o fenotipo Mutador Previo. La inactivación permanente de un mutador primario como el hMLH1 presente desde el nacimiento, produce una inestabilidad que conduce a mutaciones secundarias en la vía Mutadora, como las presentes en los genes hMSH3 y hMSH6, lo cual aumenta la inestabilidad genómica y acelera por lo tanto la acumulación de mutaciones en tumores. La inactivación de hMLH1 parece que se produce por hipermetilación del promotor del gen, lo cual conduce al fenotipo mutador. La causa de la hipermetilación no ha sido definida, aunque se plantea la presencia de un mecanismo regulador de los patrones de metilación en todo el genoma, en el cual se produciría la mutación inicial.
Joger et al6, postulan que la naturaleza de la mutación dentro del gen también puede contribuir notablemente con el fenotipo. Ellos describieron en 5 familias danesas con CCNPH una mutación en el intrón 14 del gen hMLH1 en la región de splicing. Dichas familias presentan a su vez una historia familiar similar, donde solo se identificaron dos tipos de cáncer extracolónicos (endometrio, ampolla de Vater) comparado con otras familias que no presentan esta mutación. Miyaki3,también, se han identificado mutaciones germinales en el gen hMSH6 en pacientes que no cumplen con todos los criterios para clasificar las familias como CCNPH positivas y que presentan predominantemente cáncer de endometrio y ovario14,23.
Abstract
Around 15% of colorectal cancers (CRC) are associated with an autosomic dominant hereditary pattern; some 12.940 and 5.660 deaths register a family history of CRC. The genetic factor acquires increasing pertinence in the etiology of the disease. The notable advances in molecular biology that have taken place over the past decade led to the assertion that the loss of heterogocygity and the presence of germinal mutations in the DNA repair system are predisposing mechanisms in CRC, familiar adenomatous polyposis of the colon, caused by the first of these mechanisms, occurs in less than 1% of all cases of CRC, where as the second mechanism causes 10% to 15% of all cases of CRC.
Referencias
1. Potter J: Colorectal cancer: molecular and populations. J Natl Canc Inst 1999; 91: 916-32.
2. Cheng AJ, Jang R, Wang J, et al: Possible role of telomerase activation in the cancer predisposition of patient with hereditary nonpolyposis colorectal cancer. J Natl Canc Inst 1998; 90: 316-21.
3. Alvarado J, Otero W: Gastroenterología y Hepatología. Bogotá, Asociación Colombiana de Endoscopia Digestiva, 1996.
4. Taquin W: Implementing screening for colorectal cancer. Brit Med J 1999; 319: 1212-3.
5. Lynch H, De la Chapella A. Genetic susceptibility to non-polyposis colorectal cancer. J Med Gen 1999; 36: 801-18.
6. Lynch H, Lynch J: Genetic of colonic cancer. Digestion 1998; 59: 481-501.
7. Aaltonen L, Peltomaki P, Leach F, et al: Missense and nonsense mutation in codon 659 of MLH1 cause aberrant splicing of messenger RNA in HNPCC kindreds. Gen Chr Canc 1999; 4: 372-5.
8. Andrzej S, Wortsman J, Carison A. Colorectal cancer. In: Andrzej S, Wortsman J. Molecular of pathology of soft tissue and bone tumors. Arch Pathol Lab Med, 1999; 1246-57.
9. Schilsky R, Minsky B, Hoff P. Cancer of the colon. In: DeVita V. Hellman S, Rosenberg S. Cancer Principles & Practice of Oncology, 6ª ed, Philadelphia, Lippincott, 2000; 1216-1319.
10. Ganong W: Fisiología Médica. Editorial Manual Moderno 1992; 461-5.
11. Woon C, Stick F, Wook B, et al: Chronic alcohol consumption induces genomic but not p53-specific DNA hypomethylation in rat colon. J Nut 1999; 129: 1945-50.
12. Klung W, Cummings M: Essential of Genetics. New Jersey,Prentice-Hall 2000; 446-50.
13. Bishop M and Weinberg R: Molecular oncology. Scientific American Inc 1996; 61-84.
14. Mendelson H: The Molecular Basis of Cancer. Philadelphia Saunders Company 1995: 340-56.
15. Tsum L, Siu T, Lap P et al: Frequent microsatellite instability and mismatch repair gene mutations in young chinese patients with colorectal cancer. J Natl Canc Inst 1999; 91: 1221-6.
16. Loukola A: De la Chapelle A and Aaltonen L. Strategies for screening for hereditary non-polyposis colorectal cancer. J Mol Gen 1999; 36: 819-22.
17. Lynch H: Hereditary non-polyposis colorectal cancer (HNPCC) Cytogenetics and cell genetics. Basel 1999; 86: 130-50.
18. Wooster R, Cleton J, Collins N, et al: Instability of short tandem repeats (microsatellites) in human cancer. Nat Gen 1994; 6: 152-6.
19. Shibata D, Peinado M, Ionov Y, et al. Genomic instability in repeated sequences is an early somatic event in colorectal tumorigenesis that persists after transformation. Nat Gen 1994; 6: 273-81.
20. Mecklin J, Jarvinen H, Hakkiluoto A, et al: Frequency of hereditary nonpolyposis colorectal cancer: a prospective multicenter study in Finland. Dis Colon Rect 1995; 38: 588-93.
21. Kraus C and Kastl S. A proven de novo germline mutation in HPCC. J Mol Gen 1999; 36: 919-21.
22. Aaltone L. Incidence of hereditary nonpolyposis colorectal cancer and the feasibility of molecular screening for the disease. N Engl J Med 1998; 338: 1481-7.
23. Perucho M. Cáncer del fenotipo mutador de microsatélites. Inv y Cienc 1998: 46-55.
24. Rodríguez M, Boland R, Rhamilton S, et al: A national cancer institute workshop on hereditary nonpolyposis colorectal cancer syndrome: meeting highlights and beheads guidelines. J Natl Canc Inst 1997; 89: 1758-62.
25. Juul W, Khan M, Vasen H, et al: Clinical findings with implications for genetic testing in families with clustering of colorectal cancer. N Engl J Med 1998; 339: 511-7.
26. Lindblom A, Tannergard P, Werelius B, et al: Genetic mapping of a second locus predisposing to hereditary non-polyposis colon cancer. Nat Gen 1993; 5: 279-82.
27. Espinosa A: Mecanismos de reparación de daños a la molécula del ácido. Ácido Desoxirribonucleotídico. Rev Latin Am Micr 1993; 35: 117-36.
28. Liu B, Parsons R, Hamilton S, et al: hMSH2 mutations in hereditary nonpolyposis colorectal cancer kindreds. Can Res. 1994; 54: 4590-4.
29. Nicolaides N, Papadopoulos N, Liu B, et al. Mutations of two PMS homologues in hereditary nonpolyposis colon cancer. Nat 1994; 371: 75-80.
Correspondencia:
Yenny Magnolia Montenegro Medina
Bacterióloga Lab. Genética
Universidad Surcolombiana
E-mail: yenny-mmm@yahoo.com
Neiva
* YENNY MACiNOUAMONTENEGROMEDINA. Estudiante de Maestría en Genética. Universidad de Antioquia, Medellín, Colombia. Bacterióloga Laboratorio de Genética Universidad Surcolombiana, Neiva, Colombia.
** ADONIS TUPAC RAMÍREZ CUÉLLAR, Residente 11 año de Cirugía. Universidad Surcolombiana, Neiva, Colombia.
***CARLOS MARIO MUÑH()N. Profesor de Genética, Facultad de Medicina Universidad de Antioquia, Medellín, Colombia.
